与特征选择类似,我们可以使用特征提取(Feature Extraction)来减少数据集中特征的数量。其区别在于,特征选择保留原始特征,而特征提取则将数据转换或投影到新的特征空间。
本文介绍主成分分析(Principal Component Analysis, PCA),它是一种无监督的线性变换技术,主要特点是特征提取和降维。
算法思想
PCA 广泛应用于不同领域,帮助我们根据特征之间的相关性来识别数据中的模式。其原理是找到高维数据中最大方差的方向,并将其投影到与原始维数相同或更少维数的新的正交空间上。
新的子空间的正交轴(即主成分)可以被解释为在新的特征轴彼此正交的约束下的最大方差的方向,如下图所示:
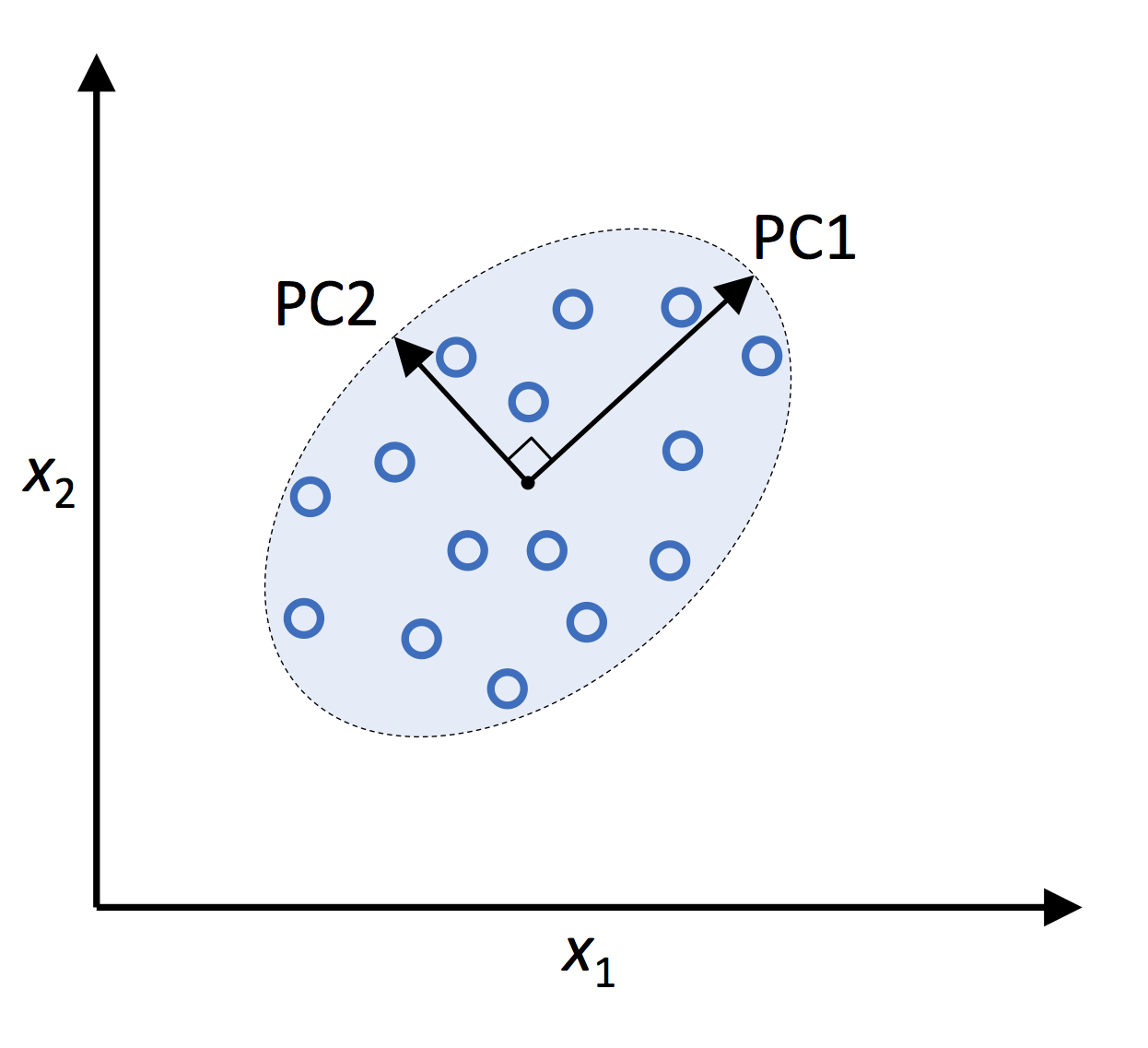
在上图中,$x_1$ 和 $x_2$ 是原始特征轴,PC1 和 PC2 是数据集的第一主成分和第二主成分。
PCA 构造了一个维度变换矩阵 $\boldsymbol W$,将高维空间向量 $\boldsymbol x$ 映射到低维空间向量 $\boldsymbol z$:
$$\begin{align} \boldsymbol x & = [x_1, x_2, \cdots, x_d], x \in R^d \\ & \downarrow \boldsymbol x \boldsymbol W, \boldsymbol W \in R^{d \times k} \\ \boldsymbol z & = [z_1, z_2, \cdots, z_k], z \in R^k \end{align}$$维度变换后,第一主成分将具有最大的方差,第二主成分将具有第二大的方差,以此类推。这些主成分必须均不相关(即使输入特征之间有相关性),数学表示为主成分向量之间两两正交。
需要注意的是,PCA 方向对数据缩放非常敏感。举例来说,假设一个特征在 [-100,100] 之间平均分布,另外一个特征在 [-1,1] 之间平均分布,PCA 会认为前一个特征重要得多。因此在运用 PCA 时,需要使用标准化将输入特征缩放到相同的尺度。
PCA 的算法流程的简单介绍如下:
- 对输入的 $d$ 维数据集 $\boldsymbol X$ 进行标准化。
- 构造协方差矩阵。
- 计算协方差矩阵的特征向量和特征值。
- 对特征值进行降序排序,对相应的特征向量进行排序。
- 提取 $k$ 个最大特征值对应的特征向量,其中 $k$ ($k \le d$) 是新特征子空间的维数。
- 使用提取的 $k$ 个特征向量构造投影矩阵 $\boldsymbol W$。
- 使用投影矩阵 $\boldsymbol W$ 对 $\boldsymbol X$ 进行变换,得到新的 $k$ 维特征子空间。
数据导入和标准化
导入 Wine 数据集 ,第 1 列为其分类标签,第 2~14 列为特征。分割数据集为 70% 的训练集和 30% 的测试集:
1 | import pandas as pd |
对训练集和测试集进行标准化:
1 | from sklearn.preprocessing import StandardScaler |
协方差矩阵的构造
接下来引入协方差矩阵。对于 $n$ 的两个特征向量(机器学习中通常用列向量表示) $\boldsymbol x_j$ 和 $\boldsymbol x_k$,其协方差如下:
$$\begin{align} \sigma_{jk} = \sigma_{kj} = {\frac 1 n} \sum_{i=1}^n (x_j^{(i)} - \mu_j)(x_k^{(i)} - \mu_k) \end{align}$$其中 $\mu_j$ 和 $\mu_k$ 分别代表 $\boldsymbol x_j$ 和 $\boldsymbol x_k$ 平均值。对于 PCA 的样本,在标准化后这两个平均值都为 0 。
协方差 $\sigma_{jk}$ 可以表示 $\boldsymbol x_j$ 和 $\boldsymbol x_k$ 的相关程度:
- 若 $\sigma_{jk}$ 大于 0,表明这两个特征正相关,即一个特征随另外一个特征的变大而变大。
- 若 $\sigma_{jk}$ 大于 0,表明这两个特征负相关,即一个特征随另外一个特征的变大而变小。
- 若 $\sigma_{jk}$ 等于 0,表明这两个特征不相关,即一个特征变化不受另外一个特征的变化影响。此时这两个特征向量两两正交。
接下来构建协方差矩阵 $\boldsymbol \Sigma$(这是大写的希腊字母 Sigma,不要与求和符号 $\sum$ 混淆)。以 3 个特征为例,其协方差矩阵如下:
$$\begin{align} \boldsymbol \Sigma = \begin{bmatrix} \sigma_1^2 & \sigma_{12} & \sigma_{13} \\ \sigma_{21} & \sigma_2^2 & \sigma_{23} \\ \sigma_{31} & \sigma_{32} & \sigma_3^2 \end{bmatrix} \end{align}$$协方差矩阵 $\boldsymbol \Sigma$ 是实对称矩阵,矩阵对角线为特征的方差,其余为特征之间的协方差。$\boldsymbol \Sigma$ 的特征向量表示主成分(即具有最大方差的方向),对应的特征值为主成分的大小。
Wine 数据集有 13 个特征,我们将从 13×13 维协方差矩阵中获得 13 个特征向量和特征值。
特征值和特征向量
求解矩阵的特征值 $\lambda$ 和特征向量 $\boldsymbol v$,使其满足条件:
$$\begin{align} {\boldsymbol \Sigma} {\boldsymbol v} = \lambda {\boldsymbol v} \end{align}$$矩阵特征值和特征向量的数学计算较为繁琐,这里不作过多介绍。
使用 numpy 内置的 linalg.eig 函数可以方便地计算特征值和特征向量:
1 | import numpy as np |
上面通过 numpy.cov 创建协方差矩阵,然后通过 numpy.linalg.eig 对其进行特征分解,产生由 13 个特征值组成的向量 eigen_vals 和 13 个特征向量(列向量)组成的矩阵 eigen_vecs。
解释方差占比
把 $d$ 维空间压缩到 $k$ 维空间,需要从中挑选包含信息最多(方差最大)的 $k$ 个特征向量(主成分)。特征值反映了特征向量的大小,需要降序排序特征值,然后提取前 $k$ 个特征值对应的特征向量。
在提取特征之前,为了观察特征的方差之间的差异,这里先对特征值的解释方差占比(explained variance ratio)进行绘图:
1 | import matplotlib.pyplot as plt |
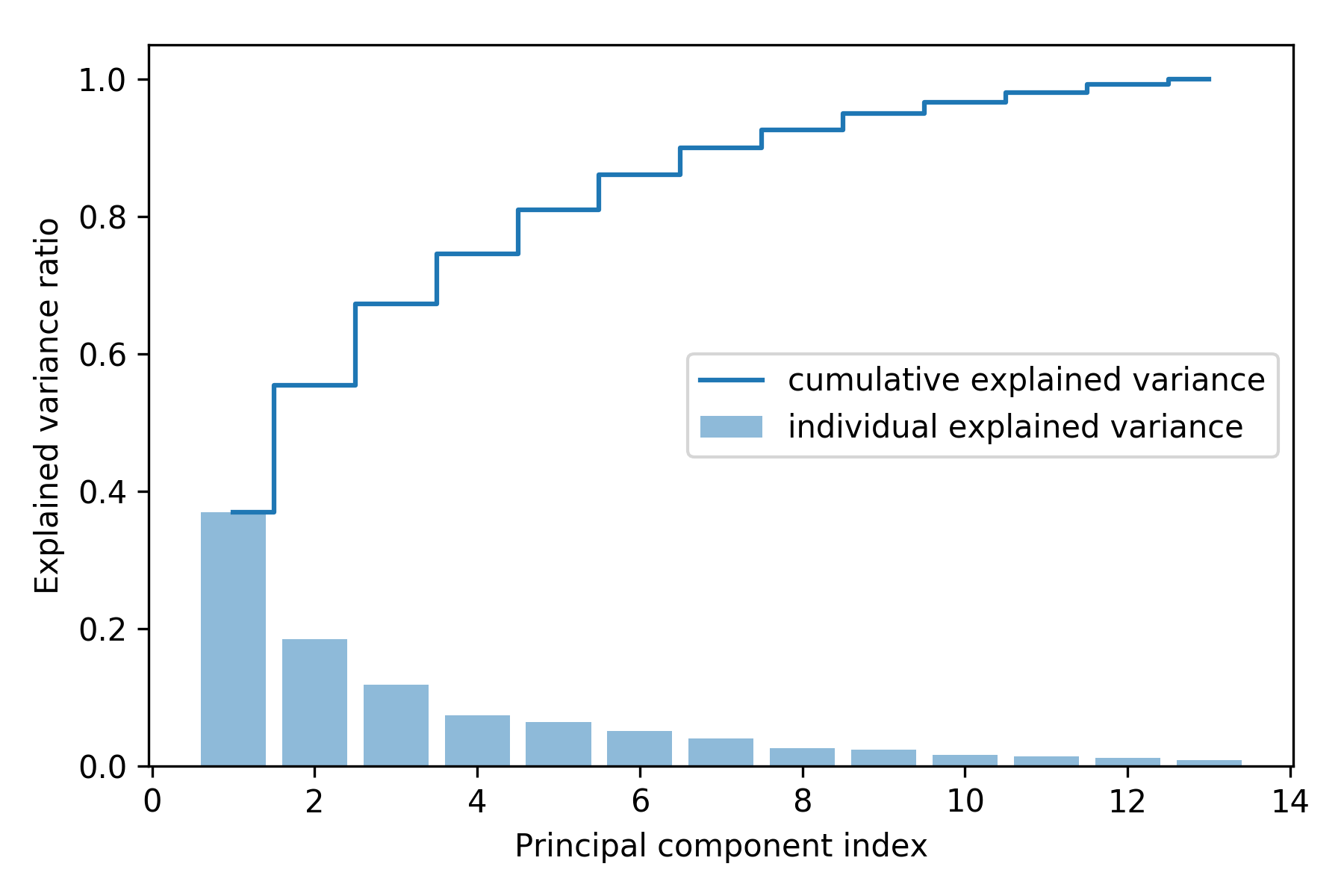
可以看到,第一主成分大约占了方差的 40%,第二主成分大约占了总方差的 20%,这两个特征值解释了接近 60% 的数据集方差。
需要注意的是,整个过程中没有使用 y_train。PCA 属于无监督学习,在分析和提取主成分的过程中从来不使用类的标签。
特征提取
本节以 Wine 数据集进行演示特征提取的最后几个步骤:
- 提取 $k$ 个最大特征值对应的特征向量,其中 $k$ ($k \le d$) 是新特征子空间的维数。
- 使用提取的 $k$ 个特征向量构造投影矩阵 $\boldsymbol W$,
- 使用投影矩阵 $\boldsymbol W$ 对 $\boldsymbol X$ 进行变换,得到新的 $k$ 维特征子空间。
简单来说,就是按照特征值降序将特征向量进行排序,从特征向量中构造投影矩阵,并使用投影矩阵将数据转换到低维子空间。
特征向量排序:
1 | # Make a list of (eigenvalue, eigenvector) tuples |
以两个主成分为例,构造投影矩阵:
注意,为了后面的绘制二维散点图,因此这里只挑选了两个主成分。实际中应该根据效率性能的平衡来确定主成分的个数。
1 | w = np.hstack((eigen_pairs[0][1][:, np.newaxis], eigen_pairs[1][1][:, np.newaxis])) |
使用投影矩阵转换原始数据集:
$$\begin{align} \boldsymbol x' & = \boldsymbol x \boldsymbol W \\ & \downarrow \\ \boldsymbol X' & = \boldsymbol X \boldsymbol W \end{align}$$其中 $\boldsymbol X$ 为原始数据集,$\boldsymbol x$ 为其中的单个样本;$\boldsymbol X’$ 为变换后的数据集, $\boldsymbol x’$ 为其中的单个样本。
1 | X_train_pca = X_train_std.dot(w) |
原始的 Wine 数据集有 13 维,通过特征矩阵 $\boldsymbol W \in R^{13 \times 2}$ 后降为 2 维,对降维后的数据集进行绘图:
1 | colors = ['r', 'b', 'g'] |
从下图可以看到,数据沿着 $x$ 轴(第一主成分)比沿着 $y$ 轴(第二主成分)更加分散,与上节中第一主成分的方差较大相吻合。另外,降维后的分类数据仍保留其特征,可以用线性分类器进行分类。
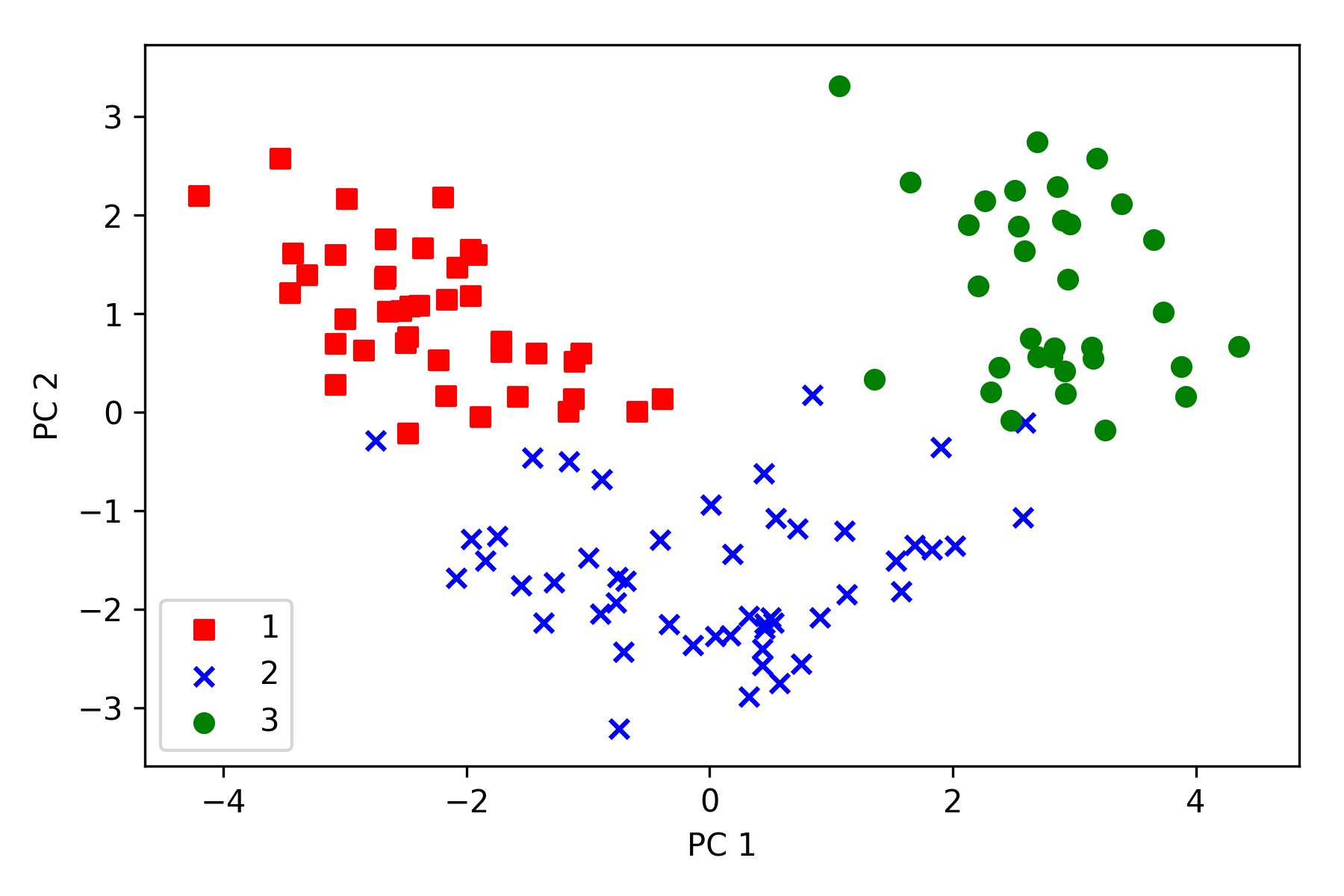
需要再次强调,PCA 是一种无监督的技术,在整个 PCA 过程中不使用任何类标签信息。
Scikit-learn 实现
边界绘图函数
plot_decision_regions见之前的文章。
通过 sklearn.decomposition.PCA 将测试集和训练集降至二维,然后使用逻辑回归对降维后的测试集进行线性分类:
1 | from sklearn.linear_model import LogisticRegression |
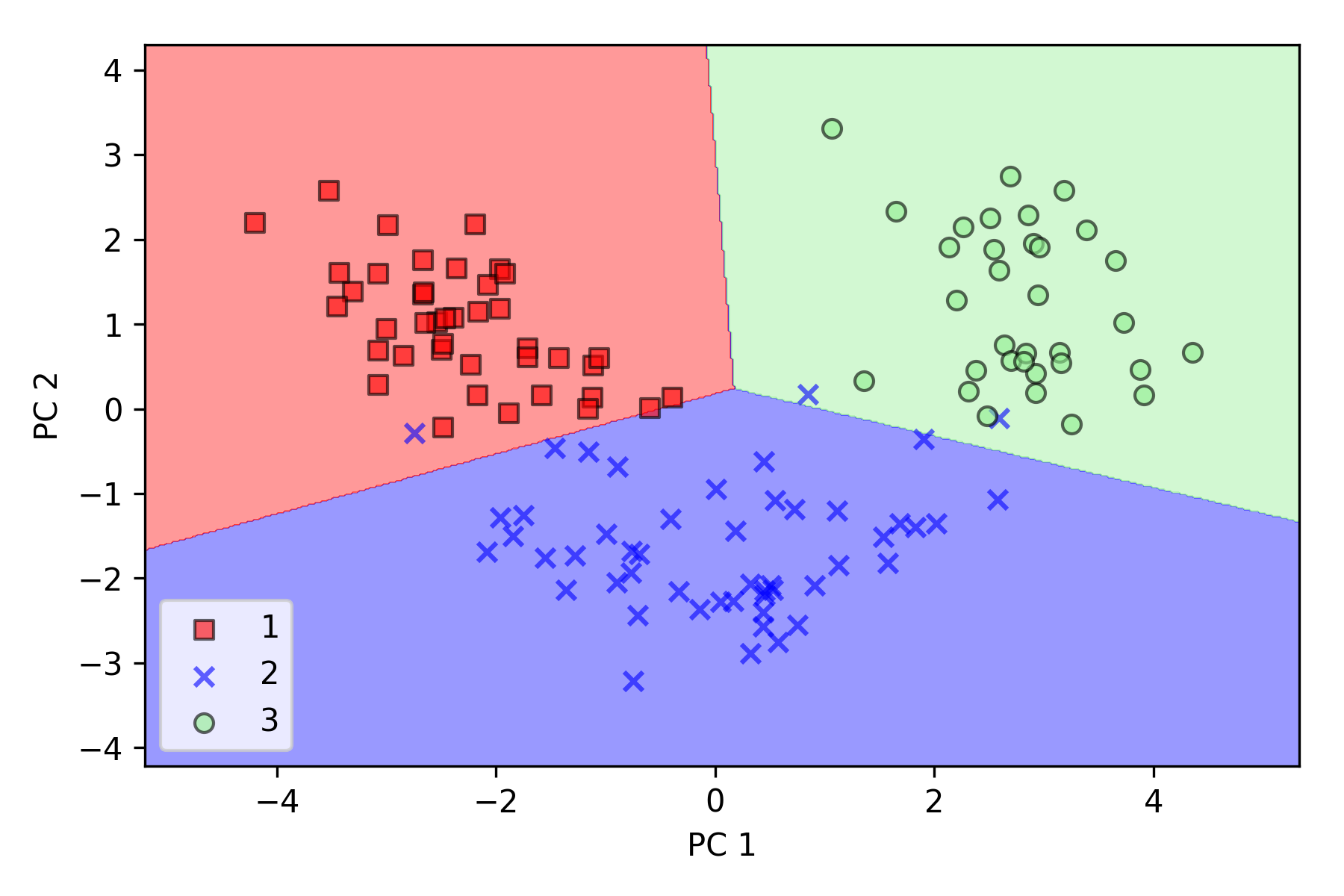
在降维空间绘制已通过训练得到的决策边界,观察是否能够进行很好地分类:
1 | plot_decision_regions(X_test_pca, y_test, classifier=lr) |
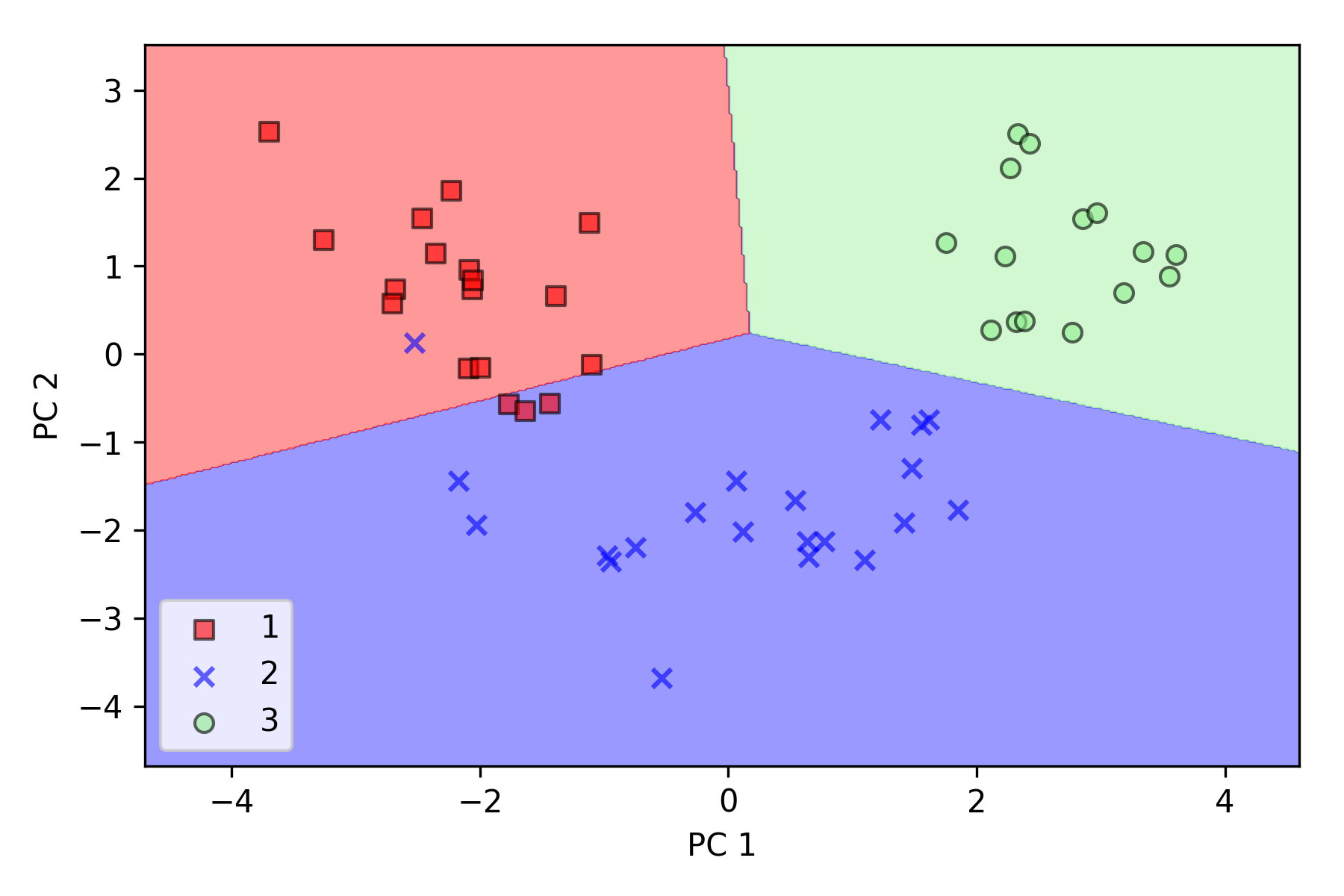
可以看到逻辑回归在新的二维子空间上表现很好,在测试数据集上只有很少样本分类错误。
关于数据集的解释方差占比,可以设置 PCA 的参数 n_components 为 None,对测试集进行变换后通过 explained_variance_ratio_ 属性得到:
1 | pca = PCA(n_components=None) |